Aggiornare la mappatura delle mutazioni genetiche per ampliare la lista di quelle che potrebbero avere conseguenze sulla salute umana. E’ il compito del software sviluppato dai ricercatori dell’Università di Firenze, in grado di identificare la presenza di mutazioni in posizioni del Dna che fino a oggi non erano state riconosciute.
Lo studio coordinato da Alberto Magi, ricercatore del Dipartimento di Medicina sperimentale e clinica, e da Gian Franco Gensini, ordinario di Medicina interna, dell’Ateneo fiorentino, è stato pubblicato sull’ultimo numero della rivista scientifica BMC Genomics (“Characterization and identification of hidden rare variants in the human genome”, DOI: 10.1186/s12864-015-1481-9).
“Dal 2003, quando il progetto genoma umano (HGP, acronimo di Human Genome Project) ha rilasciato la prima versione completa della sequenza delle basi azotate che formano il DNA, tale mappa è diventata il punto di riferimento per lo studio genetico delle malattie ereditarie e complesse. - spiega Alberto Magi - Tuttora, per identificare le varianti genetiche, le tecnologie di sequenziamento di nuova generazione continuano a paragonare il genoma di un individuo con quello di riferimento HGP.
“Elaborando i dati di migliaia di genomi di individui considerati sani rispetto al genoma di riferimento, sequenziati da vari consorzi di ricerca internazionali, - spiega Magi - abbiamo scoperto l’esistenza di oltre 100.000 errori nella sequenza prodotta da HGP che gli attuali metodi di sequenziamento non avevano individuato: in altrettante posizioni del DNA, la base azotata non coincide con quella presente nei soggetti analizzati.
Ciò si deve - prosegue il ricercatore - principalmente a errori di sequenziamento e a mutazioni presenti nel genoma dei donatori utilizzati a suo tempo dal progetto genoma umano. Per intendersi - commenta Magi - si tratta di circa 10.000 possibili mutazioni potenzialmente pericolose per la salute umana, in particolare patologie neurologiche, tumori e malattie cardiovascolari.”
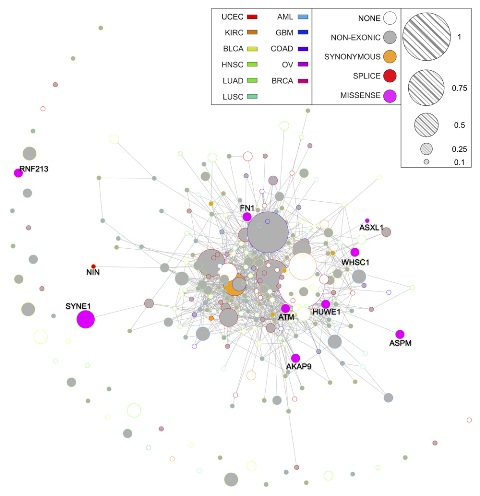
Per correggere gli errori del genoma di riferimento, il gruppo guidato da Magi -composto da Betti Giusti e Rossana Abbate, dell’Ateneo fiorentino e Azienda ospedaliero-universitaria di Careggi, e dai colleghi dell’Università di Bologna, del CNR di Pisa e del CNR di Milano - ha sviluppato un software che consente di identificare la presenza delle mutazioni genetiche. Il software è stato testato per analizzare il sequenziamento di 15 individui affetti da melanoma uveale e ha identificato più di 1500 mutazioni, correggendo il tiro delle altre rilevazioni statistiche.
“I risultati ottenuti grazie al nostro software potranno contribuire ad aggiornare la mappa delle mutazioni genetiche a disposizione della comunità scientifica. - conclude Magi - L’inclusione di queste mutazioni nelle analisi genetiche può cambiare notevolmente l’identificazione dei geni responsabili o associati a malattie ereditarie complesse.”
Fonte: Università degli Studi di Firenze